天然药物反向寻靶系统
《天然小分子化合物反向寻靶报告》
1 目的
针对客户提供的1个化合物进行反向虚拟筛选,结果返回给客户。
2 材料与方法
2.1 反向虚拟筛选材料
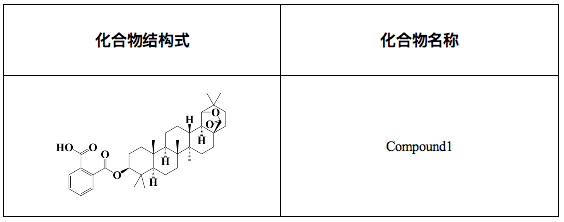
客户提供的1个化合物见表 1。
表1.客户提供的五个化合物
2.2 反向虚拟筛选流程
对接软件:Glide或者Vina软件。
方法:基于受体的反向虚拟筛选方法基于分子对接原理,将小分子化合物一一对接到靶标数据库的靶标活性位点上,根据化合物-靶标的结合能大小打分排序,结合能越低,排名越前,潜在靶标的可能性越大。
蛋白对接筛选库:我们针对Scpdb数据库,并结合我们自行研发的反向虚拟筛选数据库,利用对接软件进行反向虚拟对接筛选,蛋白质数据库共计23456种类蛋白质。该蛋白质数据库整合了目前RCSB PDB数据库中所有已知晶体蛋白结构类型数据,具有最为全面的蛋白结构数据信息。对1个化合物分别提交反向虚拟筛选任务并返回前500个分子对接结果。 |
3 结果与分析
每个化合物结果文件包括得分排名前500 对接文件信息,对接得分排序的前500个结果见列表Top_Target_500.csv 文件,我们挑选其中前50名小分子进行重点分析,如下表所示:
挑选能量最好、聚类最多的小分子位相用于结果分析。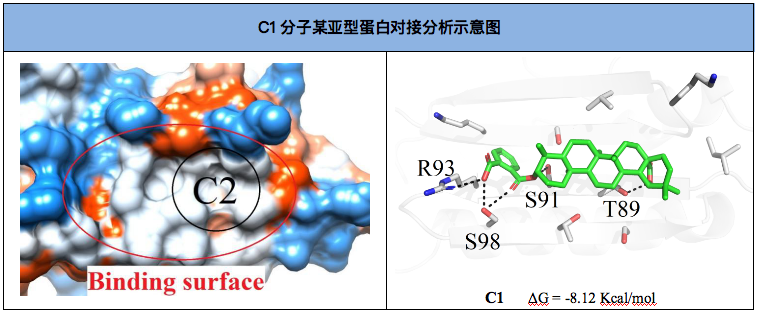
以 compound1-Whole库反向虚拟筛选排名第一的靶标结果为例,简要介绍如何通过靶标信息分析和结合模式分析来查看反向虚拟筛选结果。compound1 排名第一的靶标为Cytochrome P450 11B2, mitochondrial[Homo sapiens](第一列),靶标蛋白质文件为4dvq_1CA_10_[J](第三列,意思是PDBID:4dvq,结晶结构配体名称1CA,chain ID:J链)。在筛选时,如果筛选的目标蛋白为人源蛋白,非筛选的蛋白为其他物种来源,则需要考虑其他物种来源蛋白与人员蛋白的序列同源性,否则可以直接放弃此靶标。
3.2.1 靶标信息分析
Compound1 排名第一的靶标为 cyclin-dependent kinase 2 isoform 1 [Homo sapiens], 靶标相关信息在Top_Target_500.csv 文件,详细介绍可参考帮助文档 http://reversedock.vslead.com/index.php?r=site/help。通过对已知化合物的药理活性、治疗领域的了解,结合 Top_Target_500.csv文件中的靶标UNP_AC(第四列)数据,去对应UniProt数据库搜索更全面靶标信息,最终确定化合物的潜在作用靶标。
3.2.2 化合物和靶标蛋白结合模式分析
compound1排名第一的结果文件为4dvq_10_1CA.pdbqt 和4dvq_10_1CA.log。4dvq_10_1CA.log为化合物对接后构象得分。分析4dvq_10_1CA.pdbqt化合物对接后构象,可用可视化软件(如 Pymol,如Pymol打开有问题显示化合物键有问题,可用Discovery Studio)打开,结合蛋白质三维结构(此处为 PDBID: 4dvq,在http://www.rcsb.org/下载即可),分析化合物-蛋白质结合模式(图2)和静电表面结合情况(图3)
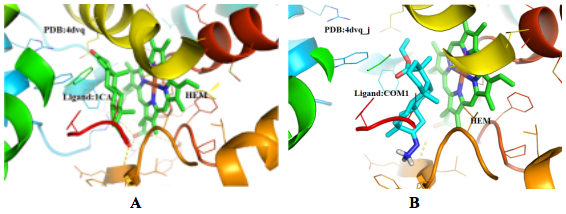
图2 结合模式图 A) 晶体结构pdb:4dvq 结合模式图; B)compound1 与 pdb:4dvq结合模式图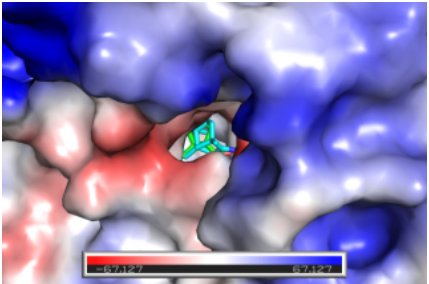
图3静电表面图晶体结PDB:4DVQ ,棍状化合物分别为配体1CA(green)和COM1(cartoon)。
该部分为前期数据结束以后,客户最广泛要做的业务,在此提供仅供参考
4 基于鉴定蛋白的药物分子设计与化学修饰
计算机辅助药物设计
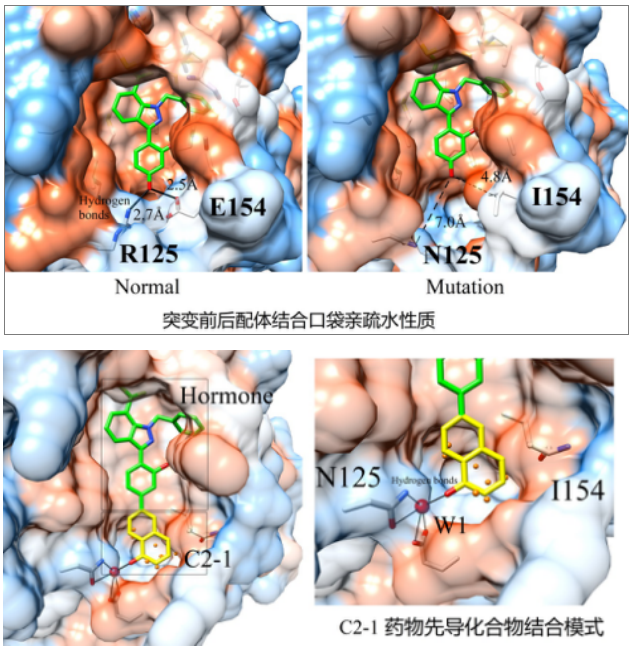
在鉴定所得蛋白质三维结构基础上,彻底分析了XYZ蛋白结合口袋周围理化性质,报告如下:(a)突变前:小分子包围在疏水口袋,亲水氨基酸R125、E154与其形成氢键铆钉作用,使其牢牢稳固在蛋白质中;(a)突变后:疏水环境大致不变,但氨基酸N125、I154造成很大空隙,亲水性转换为疏水口袋,稳定性降低。
分子动力学方法
利用分子动力学方法对XYZ蛋白突变后与小分子Hormone间相互作用状况进行了计算模拟分析,以期得到蛋白质在突变后的小分子结合信息。
(a)突变后XYZ蛋白Region A区域没有变化,对该部分不做任何修改; (b)Region B区域中,小分子下方出现巨大空洞,需要对该部分进行补漏; (c)Region B区域巨大空袭,在小分子稳定结合时,下方出现由10~13个水分子组成的“水氢键网络体系”; (d)“水氢键网络体系”中,水分子能量Energy(球体大小)和占有率Occupancy(球体颜色)均能表达出蛋白质局部区域理化性质。 具体总结为:Region B区域绝大多数水分子能量Energy极小、占有率Occupancy较小,说明该区域极为疏水;但其中W1水分子,能量较好、占有率较高,说明W1水分子极为稳定,周围氨基酸为极亲水性氨基酸,将来可以通过替换W1水分子或通过桥键作用对其进行化学修饰 |
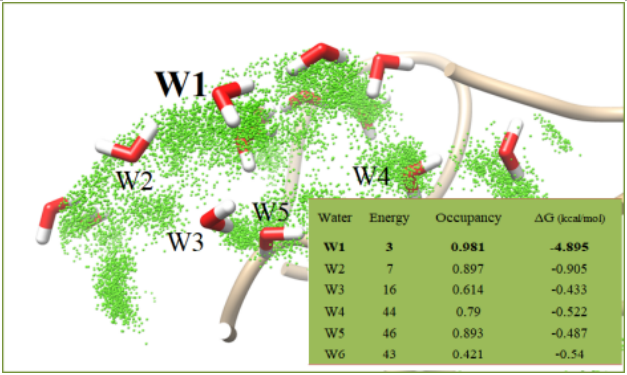

基于体内原生激素Hormone的药物设计
化学片段增长设计法
基于分子动力学模拟结果,基于W1水分子相关信息,对Region B区域进行了化学片段结构设计。首先设计的化学片段,将能够有效的到达W1水分子附近,或将其替换掉。共设计母核结构50枚,且均具备有机合成实验方法实现的可能性。
分子对接方法判定最佳化学母核结构
利用分子对接方法,将50枚母核结构分别对接进入突变后蛋白质XYZ结构中,利用分子对接权重值,判定3枚小分子药物在对接结果中占据较好的权重位置。采用C2、C1和C3进行后期化学结构改造。
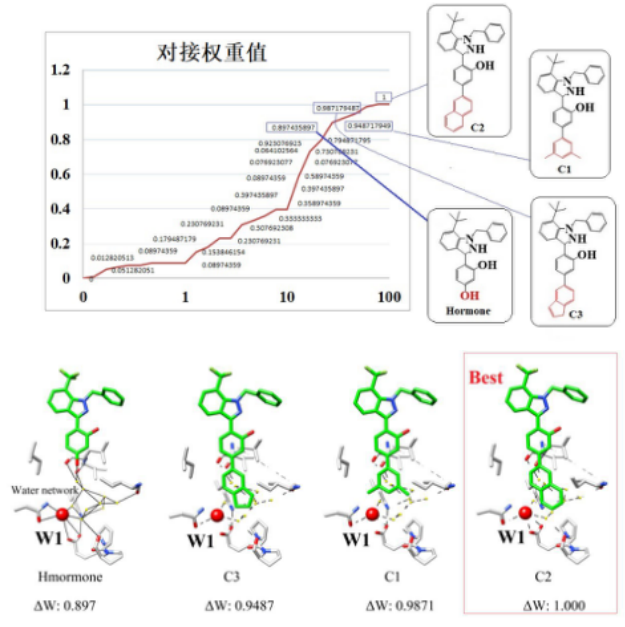
新型母核结构修饰,分子再对接
对C2~3三类化合物进行化学片段化结构修饰。尤其以第2类C2化合物为重点研究对象。基于C2化合物,共设计化学片段78枚,而后再对这78枚新型化学分子进行Molecular docking操作,预测其结构属性。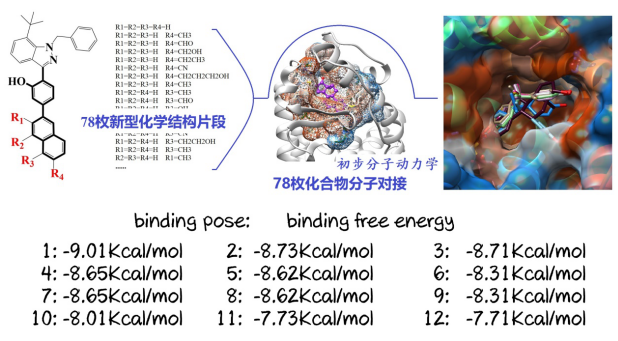
通过分子动力学模拟,计算蛋白质Protein与小分子Ligand之间的能量体系,将最有能量体系进行排名,得到前50位较好能量体系的小分子。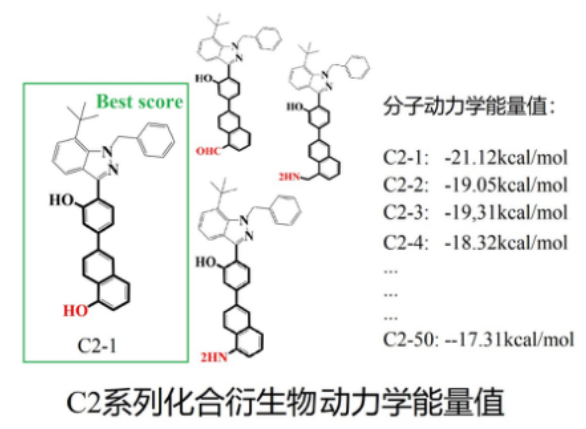
化学合成
得到化合物结构后,进行化学合成工作。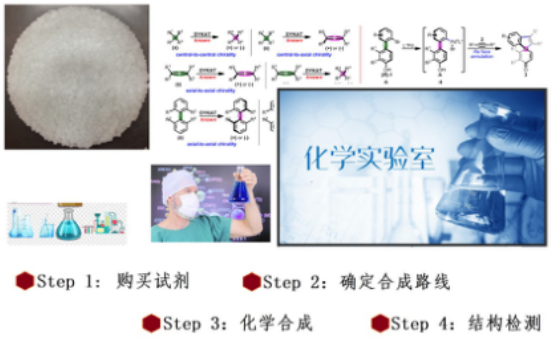
体外细胞活性实验
(1)肿瘤细胞培养
(2)细胞活性实验
细胞毒性、细胞迁移、活细胞成像、实时动态活细胞成像、非标记活细胞成像。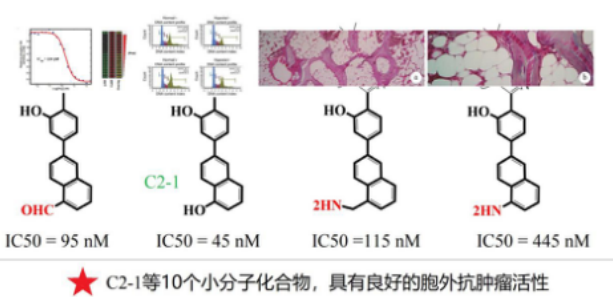
非常好的结果是:活性实验良好,特别是C2-1等10个化合物对癌细胞的细胞活性数据IC50=45nm,具备极高的成药潜在性。
动物肿瘤模型构建
成立生物活性实验室,加速动物活体实验。
将乳腺癌肿瘤细胞系,稀释注射进入小白鼠、大白鼠、白兔和黑猩猩体内,成功得到动物体内肿瘤模型。
动物体内活性实验

